L’alphabet génétique s’agrandit
L’alphabet génétique s’agrandit
Par Donovan Thiebaud
Une équipe internationale est parvenue à enrichir le patrimoine génétique de bactéries en ajoutant deux bases artificielles supplémentaires aux bases A, T, G et C sélectionnées par l’évolution pour constituer l’ADN.
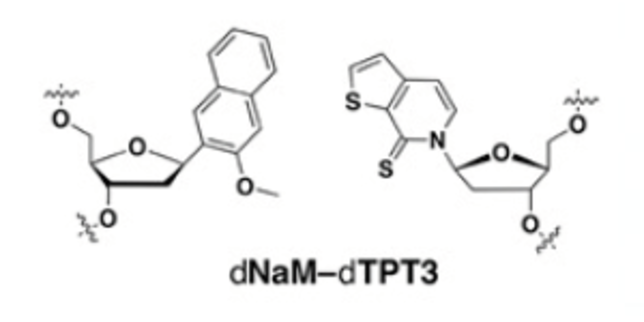
Formule topologique de la nouvelle paire de bases introduites dans des bactéries « E. coli ». | http://www.pnas.org/cgi/doi/10.1073/pnas.1616443114
A, T, G et C. Ce sont les quatre bases azotées qui, reliées deux à deux, constituent les barreaux de la double hélice de l’ADN. Tous les lycéens les retiennent par cœur depuis plusieurs générations : A pour adénine, T pour thymine, G pour guanine et enfin C pour cytosine. Elles servent principalement au stockage, à l’expression et à la transmission de l’information génétique.
Mais quatre bases, c’est trop peu pour l’équipe de Floyd E. Romesberg. Ces chercheurs ont réussi à élaborer deux nouvelles bases, baptisées X et Y, mais surtout à assurer leur maintien lors de la réplication de l’ADN in vivo. Le but : créer des organismes semi-synthétiques, avec une information génétique enrichie (3 paires de bases au lieu de 2), afin de les doter de fonctions inédites.
« Le 13e travail d’Hercule »
Pour arriver à ce résultat, l’équipe a d’abord dû choisir les bases X et Y parmi un certain nombre de candidats. Les deux bases devaient être parfaitement complémentaires, comme le sont A et T d’une part, et G et C d’autre part, afin que la réplication puisse se faire sans aucun souci. Un travail d’une quinzaine d’années. Philippe Marlière, directeur scientifique à l’Institute of Systems and Synthetic Biology (ISSB), est admiratif du travail fourni : « Ils sont partis de rien. Ils ont fabriqué 60 candidats, donc 3 600 paires possibles et ont réussi à en sélectionner une, puis à l’optimiser. Pour moi, c’est le treizième travail d’Hercule ! » Les deux bases sélectionnées ont été les composés NaM et TPT3.
Une fois ces deux bases identifiées, le principal défi consistait à assurer leur réplication une fois insérée dans un organisme – en l’occurrence, la bactérie E. coli. Un plasmide, c’est-à-dire une molécule d’ADN circulaire fabriqué in vitro, contenant une paire de bases X et Y, a été importé dans la bactérie.
Dans un second temps, des bases X et Y libres ont été ajoutées dans le milieu de culture et le génome de la bactérie a été modifié pour produire une protéine permettant le passage de ces bases libres vers l’intérieur de la bactérie. Une fois à l’intérieur, elles ont été prises en charge par les enzymes qui assurent normalement la réplication de l’ADN d’E. coli et qui placent, en face de chaque base azotée, sa base complémentaire. Chaque brin du plasmide a ainsi été efficacement copié, la séquence AGCXT devenant TCGYA.
Mais la réplication de X et de Y n’était pas parfaite, explique Thomas Lavergne, chercheur CNRS au département de chimie moléculaire de l’Université Grenoble-Alpes, qui fait partie de l’équipe : « Dans certains cas, aucune base n’était placée en face de X ou de Y ou alors il y avait une mauvaise base, A en face de X par exemple. Cela entraînait alors une mutation de l’ADN et non une réplication. Il nous fallait trouver une solution, car la fidélité n’était pas satisfaisante. »
Cette solution s’appelle CRISPR-Cas9. Cette enzyme au nom barbare permet en effet de couper l’ADN à un endroit souhaité. L’équipe de Floyd E. Romesberg a utilisé ce dispositif pour couper les morceaux d’ADN qui avaient muté. Pour Philippe Marlière, « l’utilisation de CRISPR-Cas9, en plus d’être une technique très élégante, a aussi été très efficace. Cela a considérablement amélioré la rétention de la paire de nucléotides X et Y. La réplication était bien meilleure ».
Influence de CRISPR-Cas9 sur la rétention de la paire de bases azotées X et Y après 3 réplications de l’ADN. Les 16 différents cas correspondent à la position de la base X dans l’ADN. Pour chacun des cas, la rétention est affichée avec ou sans l’action de CRISPR-Cas9. Avec l’action de Cas9, pour 14 des 16 cas, la rétention, et donc la stabilité de l’organisme est très satisfaisante. | http://www.pnas.org/cgi/doi/10.1073/pnas.1616443114
Perspectives médicales ?
Fort de ce résultat encourageant, l’équipe a ensuite testé cette technique de réplication de l’ADN modifiée d’E. Coli dans des conditions plus difficiles et dans différents milieux, liquides ou solides. Les résultats sont restés très satisfaisants. Au fil des réplications, la perte d’information par suppression de bases azotées ou par mutation a été négligeable et la croissance des bactéries n’a été ralentie que de 10 %, un chiffre remarquable. « Nous avons même réussi à assurer une bonne réplication en insérant deux paires de bases X et Y, au départ dans la bactérie, au lieu d’une seule », se félicite Thomas Lavergne, avant d’ajouter : « C’est important pour la suite car nous voulons pouvoir modifier plus largement l’ADN des organismes et donc placer plusieurs paires de bases X et Y à des endroits différents. »
Les chercheurs ne veulent pas s’arrêter là. La réplication ADN-ADN avec ces deux nouvelles bases n’est qu’une première étape. La prochaine, c’est le passage de l’ADN à l’ARN afin par la suite de produire des protéines qui seraient constituées d’aminoacides modifiés par l’homme. « Il serait ainsi possible de fabriquer des protéines thérapeutiques totalement nouvelles. Cela ouvrirait des perspectives médicales extrêmement importantes. De plus, nous voulons aussi étendre cette technique à d’autres organismes qu’E. coli, notamment aux cellules eucaryotes », précise Thomas Lavergne.
Le chercheur tient également à rassurer sur cette découverte : « Nous avons montré qu’en l’absence des bases X et Y dans le milieu de culture, l’ADN semi-synthétique contenant par exemple ATGXC était rapidement converti en ADN naturel ATGAC. Ces bases X et Y étant exclusivement synthétiques et absentes de notre environnement, la propagation de ces organismes hors des frontières du laboratoire est donc impossible. »
Le bioconfinement est donc assuré dans cette étude aux résultats prometteurs qui n’est en fait que le début d’une nouvelle ère au sein d’une discipline en pleine expansion, la biologie de synthèse.








